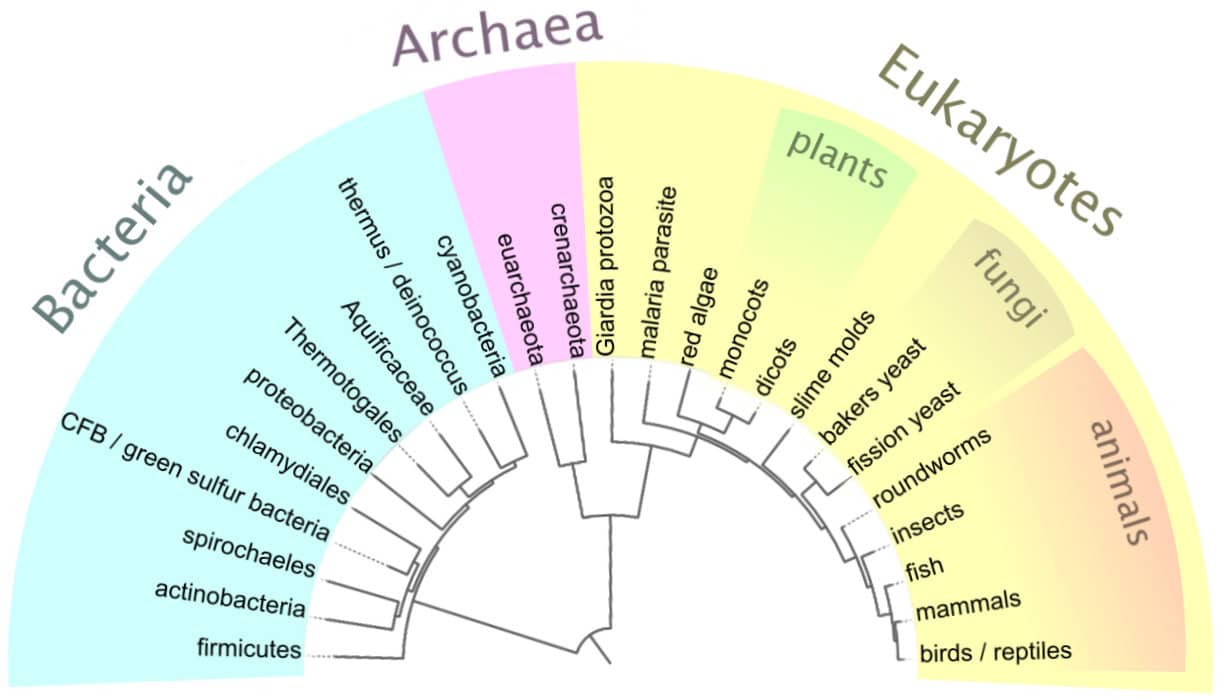
Les Arbres Phylogénétiques Évolutionnistes sont-ils corrects?
Les arbres évolutionnaires (aussi appelés arbres phylogénétiques) sont des représentations graphiques qui illustrent les relations supposées de parenté évolutive entre différents organismes, espèces, ou groupes taxonomiques. Ils sont un outil central dans la biologie évolutive pour visualiser comment les scientifiques pensent que la diversité du vivant s’est développée à partir d’ancêtres communs.
Un arbre évolutionnaire est un diagramme en forme d’arbre:
- Les branches représentent des lignées d’organismes.
- Les nœuds (points de bifurcation) représentent des ancêtres communs hypothétiques.
- Les feuilles (ou extrémités des branches) représentent les espèces vivantes ou fossiles observées aujourd’hui.
Le modèle darwinien en danger
Il y a un nombre croissant de voix scientifiques (souvent évolutionnistes eux-mêmes) qui reconnaissent les limites, contradictions et insuffisances de la théorie actuelle (modèle néo-darwinien), tout en appelant à un renouvellement profond du paradigme.
Il y a des contradictions entre arbres morphologiques et moléculaires. Une étude de l’Université de Bath (2022) intitulée « Most of our evolutionary trees could be wrong » montre que les arbres phylogénétiques basés sur l’anatomie (forme, structure) contredisent souvent ceux construits avec les données moléculaires (ADN).

Inscrivez vous sur QQLV!
Pour soutenir l’effort du ministère et la création de contenus:
Oyston, J.W., Wilkinson, M., Ruta, M., and Wills, M.A., Molecular phylogenies map to biogeography better than morphological ones, Communications Biology 5:521, 31 May 2022.
- Exemple avec les Afrotheria (éléphants, taupes dorées, lamantins…) qui sont très différents morphologiquement, mais semblables génétiquement.



Cela remet en question le postulat que la ressemblance morphologique = parenté évolutive. Les arbres moléculaires reflètent souvent des proximités géographiques, non des ressemblances anatomiques. Des espèces très différentes mais vivant en Afrique sont groupées ensemble par l’ADN. Cela affaiblit l’idée que la sélection naturelle à partir de mutations aléatoires est suffisante pour expliquer les lignées de vie.
Dans The Guardian (2022) on lit:
« Le récit classique de l’évolution dans les manuels est absurde, simpliste et trompeur. »
Dans Brown & Hullender (2022) on lit:
« Les mutations aléatoires + sélection naturelle ne peuvent pas produire des nouvelles structures complexes.
La probabilité de l’évolution darwinienne est si faible qu’elle tombe dans le domaine de l’impossible statistique (ordre de grandeur 10⁻⁵⁰). »
Avec Fodor & Piattelli-Palmarini (2010, 2011) on lit:
« Darwin avait tort… Personne ne sait comment les phénotypes évoluent. »
Ces auteurs sont des évolutionnistes athées, donc pas suspects de biais religieux, mais pourtant très critiques.
Il y a des problèmes méthodologiques du darwinisme dénoncés comme l’homologie circulaire où on on postule que la similarité implique un ancêtre commun, puis on cite la similarité comme preuve. Il y a aussi le manque de mécanismes observables pour expliquer l’apparition d’organes complexes (problème de la complexité irréductible). Enfin la sélection naturelle semble « inactive » dans de nombreux cas de changements génétiques (Buranyi: « natural selection was asleep at the wheel »).
Les petits changements (microévolution) observables n’impliquent pas qu’on puisse expliquer les grands changements (nouveaux types corporels, organes, métabolismes). L’analogie entre les deux est abusive: on ne peut pas extrapoler indéfiniment.
James Shapiro dit que l’ADN change activement via des mécanismes intégrés (natural genetic engineering), pas par erreurs aléatoires.
Mae-Wan Ho dit que la Synthèse Moderne doit être « entièrement remplacée » ; ses défenseurs sont « ignorants en génétique moléculaire ».
Suzan Mazur, Stephen Meyer, Bob Holmes (New Scientist), tous admettent que l’apparition de nouvelles espèces reste mystérieuse.
Remise en question de l’arbre de la vie (Tree of Life)
Si les arbres phylogénétiques sont remis en question de manière sérieuse, cela a des conséquences majeures pour le modèle évolutif dans son ensemble, car ces arbres constituent une structure fondamentale de l’évolution darwinienne. L’un des postulats centraux de l’évolution est que toutes les formes de vie descendent d’un ancêtre commun unique, formant une sorte de grand arbre unique:
- Tronc = ancêtre commun universel
- Branches = grands groupes taxonomiques
- Feuilles = espèces actuelles
Si les arbres phylogénétiques sont incohérents, contradictoires ou arbitraires, alors l’idée même d’un tronc commun universel devient fragile, voire invalide. Cela suggère que la généalogie du vivant ne peut pas être retracée de manière linéaire et cohérente.
Perte de l’argument de la similarité = parenté
L’évolution repose largement sur l’idée que les ressemblances (anatomiques, génétiques, embryologiques) entre espèces sont la preuve d’une descendance commune. Mais si les arbres moléculaires (ADN) contredisent les arbres morphologiques, des espèces très différentes (ex. éléphant et taupe dorée) sont classées ensemble par génome, et des espèces morphologiquement proches sont génétiquement éloignées, alors la logique de la parenté par similarité (homologie) s’effondre. Cela enlève un pilier majeur de l’inférence évolutive.
Incapacité à retracer l’histoire évolutive
Les arbres phylogénétiques sont utilisés pour:
- reconstituer l’ordre d’apparition des espèces dans le temps,
- proposer des ancêtres communs hypothétiques,
- dater les divergences (horloge moléculaire).
Si les arbres sont instables, dépendants des méthodes, et incohérents entre eux, alors les reconstructions historiques deviennent hautement spéculatives. L’horloge moléculaire perd sa fiabilité et les difficultés s’amoncellent pour valider la macroévolution (passage entre grands groupes) sans ces arbres.
Un bon modèle scientifique doit faire des prédictions vérifiables. Or les arbres basés sur la morphologie prédisaient certaines parentés. Ces prédictions sont démenties par l’ADN (ex : les chauves-souris plus proches des chevaux que des primates). Si aucune méthode ne donne de résultats cohérents, alors le modèle ne prédit plus rien de fiable, il est faible scientifiquement.
Les arbres moléculaires révèlent souvent que les espèces proches génétiquement vivent dans les mêmes régions, même si elles sont très différentes en apparence. Cela suggère que l’environnement local ou l’écologie influence fortement le génome (via convergence ou adaptation) ce qui contredit l’idée d’un arbre fondé sur la pure lignée ancestrale.
Le modèle créationniste propose plusieurs types créés, chacun avec son arbre propre.
Si les arbres sont incertains les manuels scolaires devraient être révisés (actuellement, ils enseignent des arbres comme des faits établis). Le professeur Matthew Wills de l’Université de Bath a admis:
« … il s’avère que nous nous sommes beaucoup trompés sur nos arbres évolutifs.
Depuis plus de cent ans, nous classons les organismes selon leur apparence et leur structure anatomique,
mais les données moléculaires racontent souvent une histoire bien différente.« 1
Comment des espèces proches en apparence peuvent être éloignées génétiquement et inversement?
C’est l’un des points les plus fascinants (et déconcertants) de la biologie moderne: le paradoxe apparence vs génome.
Certaines espèces très proches génétiquement peuvent paraître très différentes.
D’autres, visiblement très proches en apparence, peuvent être génétiquement distantes.
Ce phénomène n’est pas une anomalie locale, c’est une observation récurrente, qui remet en cause l’idée simple de “plus on se ressemble, plus on est proches”. Pour comprendre cela nous pouvons utiliser l’analogie du piano:
- Le clavier du piano = le génome brut (les gènes, l’ADN)
- La partition = la régulation des gènes (quand, où et combien un gène est exprimé)
- Le jeu du musicien = les interactions complexes (environnement, épigénétique, ARN, protéines, etc.)
- La mélodie finale = le phénotype (apparence, forme, comportement, physiologie)
Deux espèces peuvent avoir des touches très similaires (un génome presque identique), mais jouer des partitions très différentes. Elles auront des formes, comportements ou structures très divergents.
À l’inverse, deux espèces peuvent jouer des morceaux très similaires (phénotypes proches), avec des claviers différents: leur génome est plus éloigné, mais le résultat visuel est trompeur.
Ce ne sont pas seulement les gènes présents, mais quels gènes sont activés, quand, dans quel tissu, et à quelle intensité. Cela dépend des mécanismes épigénétiques (méthylation, acétylation, etc.), l’ARN non codants (microARN, siRNA, etc.), les facteurs de transcription, les interactions complexes de réseaux de gènes. Ce sont ces couches de régulation qui jouent un rôle majeur dans les différences visibles.
Une grande partie du génome est partagée entre les espèces (ex. : les humains partagent 60% de leurs gènes avec une mouche). Même les petits changements dans les « interrupteurs » génétiques peuvent engendrer de grands effets visibles (ex. position des membres, taille du crâne…).
Deux espèces non apparentées peuvent avoir des traits similaires (ailes, forme du corps, etc.) parce qu’elles vivent dans un environnement similaire. D’où une ressemblance morphologique trompeuse, sans réelle parenté génétique proche. Les requins (poissons cartilagineux) et les dauphins (mammifères) ont une forme hydrodynamique très proche, mais sont très éloignés génétiquement.
Le même gène peut produire des effets différents selon le contexte (tissus, hormones, signaux extérieurs). L’embryologie montre que les mêmes gènes de développement (ex. les gènes Hox) sont présents chez beaucoup d’animaux, mais utilisés différemment ce qui produit des formes très variées.
Les divergences morpho/géno peuvent aussi interroger la baraminologie (classification créationniste) bien que l’un de ces critères essentiels soit l’hybridation, lorsque les organismes peuvent se reproduire ensemble et signalent donc l’appartenance à un même genre (non testable sur les fossiles anciens cependant).
La baraminologie ne considère pas la génétique brute comme définitive. Pourquoi?
- Des gènes identiques peuvent être partagés sans lien de parenté directe (réutilisation par design).
- Des similitudes génétiques peuvent refléter des pressions écologiques similaires (design convergent).
- Certains outils bioinformatiques peuvent forcer des relations génétiques via des choix méthodologiques (alignements, choix des marqueurs…).
Todd Wood et Kurt Wise ont reconnu que les arbres génétiques varient selon les gènes choisis, et donc ne peuvent être utilisés seuls pour définir un « genre ».
Par exemple l’éléphant et la taupe dorée sont proches dans l’ADN (Afrotheria), mais très éloignés dans leur apparence, écologie et comportement. Pour un baraminologiste faut-il les considérer du même « genre »? Probablement non, d’après les morphologies très divergentes. Il est peu probable que les créationnistes considèrent l’éléphant, la taupe dorée et le lamantin comme issus d’un même type créé.
Les analyses baraminologiques utilisent la discontinuité, et les Afrotheria montrent trop de discontinuité morphologique.
Un autre exemple avec le requin et le dauphin, ou le pangolin et le fourmilier. Le risque ici est de suggérer un lien créé alors qu’il s’agit probablement de convergence fonctionnelle, ce que la baraminologie ne confond pas avec un lien d’origine.
Les créationnistes ne s’attendent pas à ce que tous les gènes aient une lignée unique. On peut comprendre qu’un même gène ou même séquence se retrouve dans plusieurs baramins, sans lien de filiation. Cela correspond à l’idée de design modulaire ou réutilisé, comme un ingénieur réutilise des composants efficaces.
C’est un problème plus grave pour les évolutionnistes parce que l’évolution darwinienne repose sur l’historicité des lignées génétiques. Si les arbres moléculaires sont incohérents, des espèces très différentes morphologiquement ont des gènes très similaires, ou des espèces similaires morphologiquement sont génétiquement éloignées, alors on ne peut plus tracer une histoire évolutive claire. Cela détruit l’idée de filiation génétique fiable, compromet l’horloge moléculaire et rend les arbres phylogénétiques instables et arbitraires.
Si l’arbre s’écroule, c’est tout le cadre darwinien qui perd sa colonne vertébrale. Le modèle évolutionniste doit rendre compte d’une histoire génétique cohérente.
Le modèle créationniste n’exige pas une lignée génétique universelle, et peut interpréter les similitudes comme du design.
Dans la vision évolutionniste standard, on postule que:
- Tous les gènes présents chez les êtres vivants actuels sont issus de modifications successives d’un ancêtre commun.
- L’ADN doit donc refléter l’arbre de la vie: plus deux espèces sont proches dans l’arbre, plus leurs gènes doivent être similaires.
- On doit pouvoir reconstruire la lignée de chaque gène (et des espèces) jusqu’à l’ancêtre commun universel (LUCA).
L’ADN est supposé être le « registre fossile moléculaire ».
L’étude de Bath (2022) a montré que les arbres basés sur l’ADN contredisent ceux basés sur l’anatomie (morphologie). Les espèces regroupées par l’ADN sont souvent très différentes morphologiquement, mais proches géographiquement. Les Afrotheria (éléphant, taupe dorée, lamantin…) n’ont aucune homogénéité apparente, mais sont proches génétiquement… et géographiquement. Cela contredit l’idée que l’anatomie et les gènes reflètent toujours une lignée unique, cela remet en question l’arbre évolutionniste.
Conclusion

On peut dire avec une certaine justesse – et de plus en plus de scientifiques eux-mêmes le reconnaissent – que Darwin s’est fondamentalement trompé avec son célèbre croquis d’arbre de la vie annoté « I think ».
Ce croquis représente ce qui allait devenir le cœur conceptuel de la théorie de l’évolution: un ancêtre commun universel (le tronc) à partir duquel toute la diversité du vivant aurait émergé, par divergence progressive des lignées (les branches).
Ce croquis est devenu un symbole de la pensée darwinienne et de l’arbre de la vie. De nombreux biologistes parlent aujourd’hui de réseaux phylogénétiques:
- Transfert horizontal de gènes,
- Hybridation,
- Introgression,
- Recombinaisons ancestrales…
Autrement dit, l’histoire du vivant ne forme pas un arbre propre et ramifié, mais plutôt un verger de la vie.
Les conséquences pour la biologie évolutive et la taxonomie sont considérables. Cela complexifie:
- La classification des espèces (la morphologie seule n’est pas fiable)
- Les arbres phylogénétiques (selon qu’on regarde les gènes, les protéines, ou l’anatomie, on obtient des arbres différents)
- Les tentatives de reconstruire l’histoire évolutive
D’où l’argument créationniste et/ou intelligent design: ce que nous voyons ressemble davantage à un système modulaire, flexible et adaptable, qu’à un bricolage graduel aléatoire.
La ressemblance extérieure n’est pas un guide fiable de parenté génétique. La proximité génétique n’est pas un gage de ressemblance visible.
Le génome est comme un clavier, mais le morceau joué dépend de la partition (régulation) et du contexte (environnement, développement, plasticité).
- https://www.bath.ac.uk/announcements/study-suggests-that-most-of-our-evolutionary-trees-could-be-wrong/.

Inscrivez-vous sur QQLV!
Pour soutenir l’effort du ministère et la création de contenus:
RECEVEZ DU CONTENU par email
Recevez du contenu biblique, archéologique et scientifique dans votre boîte mail!